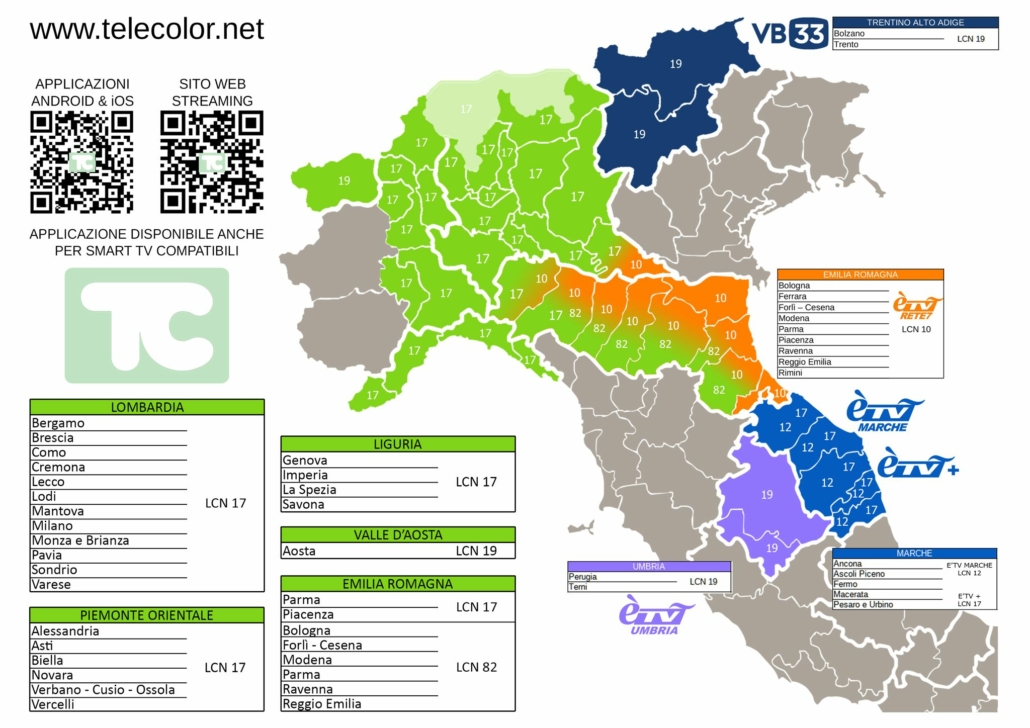
Durante il Montalcini Global Biotech Forum di Doha, La Società Educazionale di Medicina di Precisione presenterà terapie all’avanguardia per il trattamento di malattie metaboliche e infiammatorie associate all’invecchiamento, con un ‘attenzione particolare alla ricerca sia pre-clinica che clinica. L’obiettivo è esplorare lo sviluppo industriale di nuovi approcci terapeutici per le malattie croniche non trasmissibili (MCNT), una delle principali sfide sanitarie del XXI secolo.
The Educational Society of Precision Medicine (S.I.E.M.PRE.) is a non-profit scientific association dedicated to achieving national scientific and social goals. Its mission is to promote research, education, and scientific communication in the medical field through approaches rooted in “Personalized Precision and Gender Medicine.”
Precision Medicine is an innovative approach that provides accurate predictive and preventive diagnoses, along with safe and effective therapies. It takes into account the genetic, physical, emotional, holistic characteristics, and environmental factors of each patient, enabling tailored diagnoses and therapeutic strategies based on individual responses to treatments. This is made possible through the use of pharmacogenetic analyses and tools that help select the most suitable and effective medications at the appropriate doses, thereby personalizing therapeutic strategies.
S.I.E.M.PRE. operates through its scientific unit, the “Human Microbiome Care Research Institute” (Clinical Advancement in Research and Education), a national authority specializing in human microbiome research.
During the Biotech Forum in Doha, S.I.E.M.PRE. aims to discuss potential scientific cooperation and collaboration in innovative therapies for metabolic and inflammatory diseases related to aging, with a focus on pre-clinical and clinical research. The goal is to explore the industrial development of new therapeutic approaches for Non-Communicable Chronic Diseases (NCDs), one of the leading health challenges of the 21st century.
The Human Microbiome is a vital endocrine organ that interacts with the immune system, its behavior continuously shaped by genetic, epigenetic, and sex-based factors that influence disease susceptibility and response to treatment. It is integral to our immune system, capable of identifying and differentiating harmful substances, including toxic agents and tumorigenic inflammatory markers from the exposome and envirome. The Human Microbiome plays a crucial role in maintaining health, as it is involved in immune system development, the regulation of digestive and metabolic functions, and the performance of neurological functions.
Our scientific delegates and researchers make significant contributions to both National and International scientific research. They actively participate in and promote congresses, conferences, and seminars where knowledge and initiatives on the future of Precision Medicine are shared, aiming to protect medical science through global collaborations that address complex health challenges. These efforts also aim to counter anti-scientific tendencies, disease mongering, conflicts of interest, and other risk factors that do not prioritize patient or consumer well-being.